临床基因测序数据解读技巧-手把手教你如何运用SplicAI预测变异位点是否影响剪接
案例:探针:NanoWES_hg38;基因:QARS1;突变位置:chr3:49099957-49099959;经OMIM数据库查询,该基因QARS1与小头畸形、进行性、癫痫发作以及脑和小脑萎缩疾病相关;而NM_005051.3:intron14:c.1295+2_1295+4del这个变异影响到了经典剪接位点,所以,需要对该变异是否影响剪接进行再次判断;
一、案例分享-如何运用SplicAI预测变异位点是否影响剪接
案例:探针:NanoWES_hg38;基因:QARS1;突变位置:chr3:49099957-49099959;HGVS: NM_005051.3:intron14:c.1295+2_1295+4del:
经OMIM数据库查询,该基因QARS1与小头畸形、进行性、癫痫发作以及脑和小脑萎缩疾病相关;而NM_005051.3:intron14:c.1295+2_1295+4del这个变异影响到了经典剪接位点,所以,需要对该变异是否影响剪接进行再次判断;

SpliceAI分析预测具体操作步骤如下:
1.打开网址
2.输入变异:NM_005051.3:c.1295+2_1295+4del
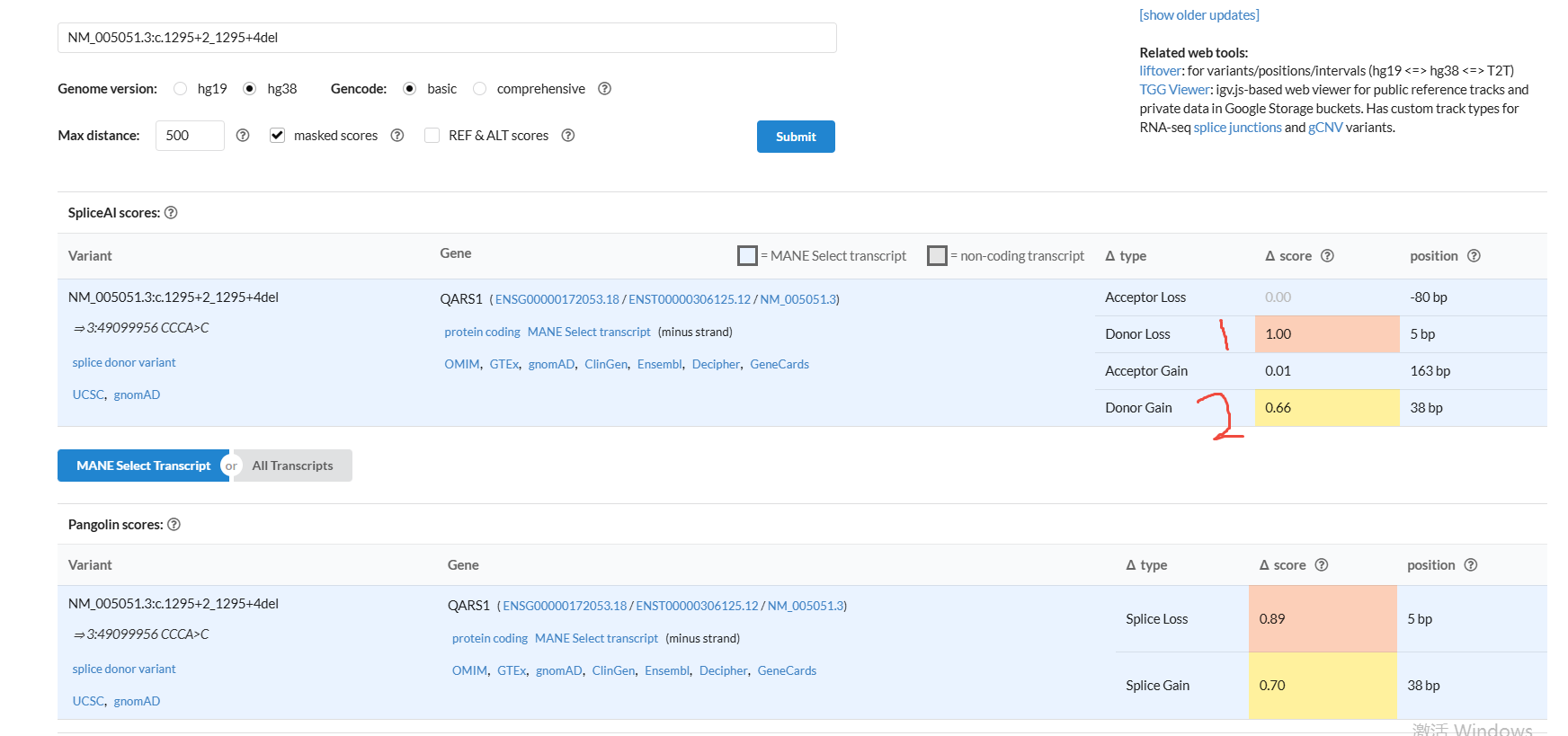
2.1.结果中的1处,Δ score =1分,代表影响剪接,position 为5 bp,代表从变异开始的位置数,往右数5 bp处会发生剪接;但是从bam图直接看,并不是在5 bp的位置发生剪接,这里和hgvs的对齐有关系;从转录本的信息3:49099956 CCCA>C可以看出来,这里具体发生的突变是CCCA>C;因此,这里的5bp是从下图红框中的chr3:49,099,955开始数的,chr3:49,099,960位置就是5 bp的位置,从SpliceAI示意图也能看出来;
bam图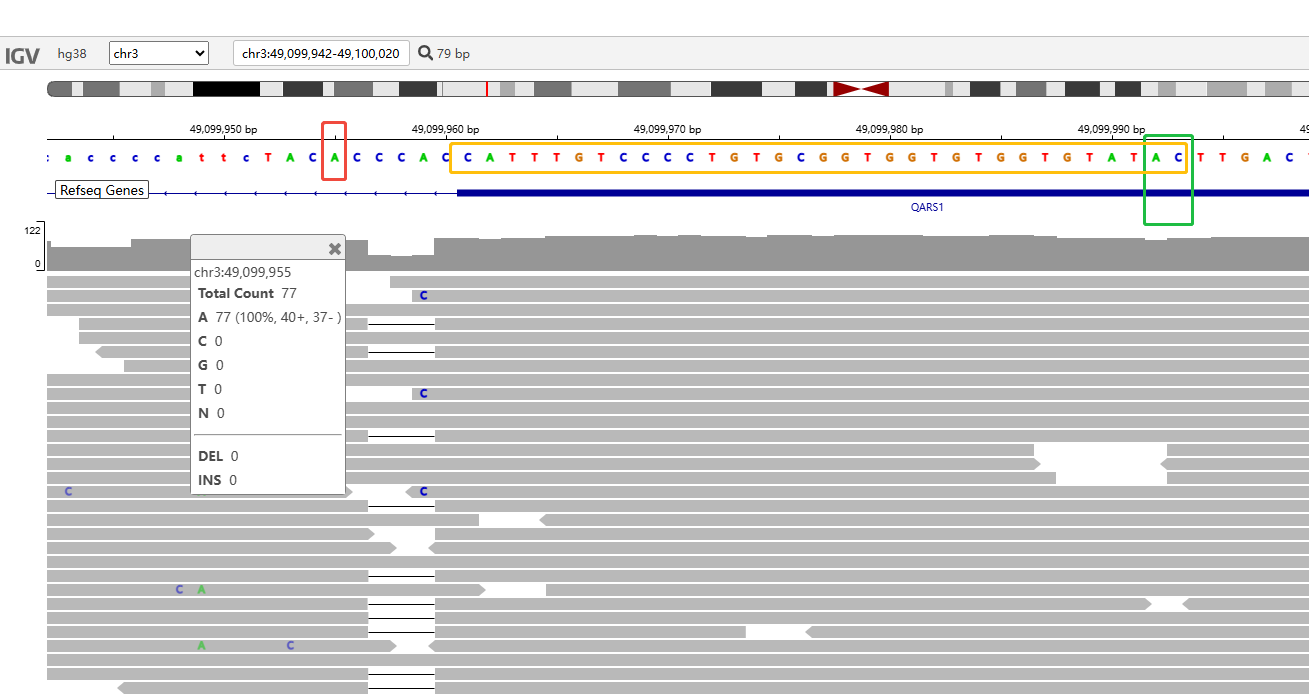
SpliceAI示意图

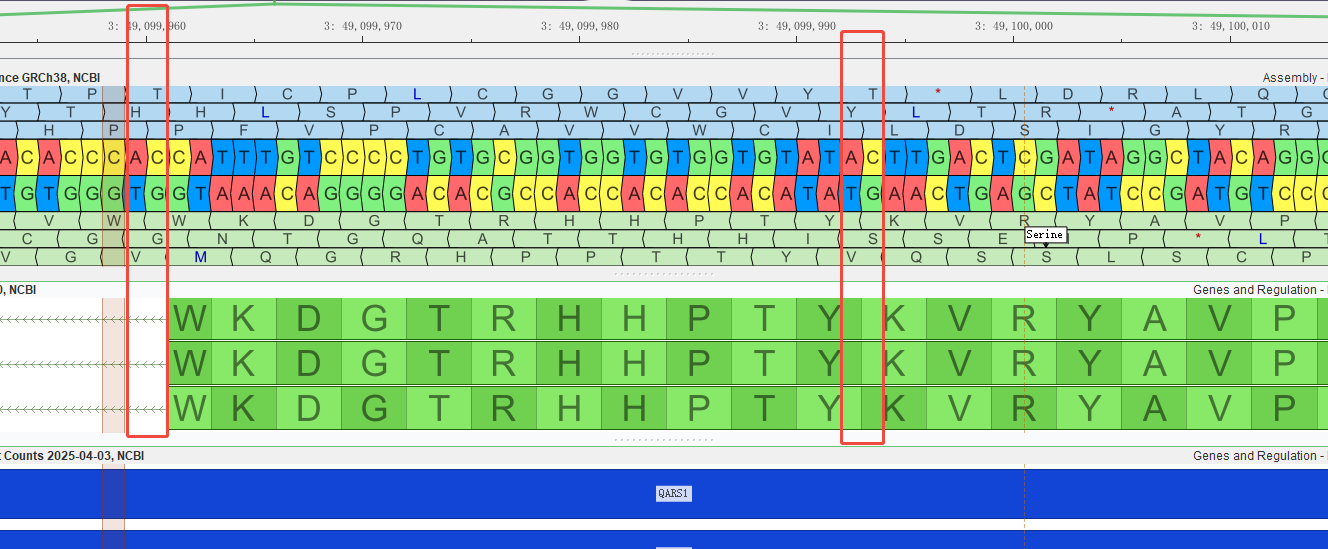
2.2. 结果中的2处,Δ score =0.66分(一般认为Δ score大于0.5或者0.2分,认为这个剪接是有效的,不同机构要求可能不一样);position 为38 bp,就在chr3:49,099,993的C处,意思就是这里的AC是可变剪接位点;如果从这个位置发生剪接的话,就需要去核查chr3:49,099,961-chr3:49,099,993这个区域是否编码蛋白,经GenomeBrowse核查,该区域是编码蛋白的,因此,发生剪接以后,chr3:49,099,961-chr3:49,099,993区域就被剪切掉了,总共是33bp,不打断阅读框,因此,这个地方的证据就需要降级使用,未打断阅读框,影响蛋白小于10%~~
GenomeBrowse核查结果
二、SpliceAI在临床数据解读中的应用
SpliceAI 是一种基于深度学习的人工智能工具,专门用于预测前体 mRNA 剪接位点的变异如何影响RNA剪接过程。在临床数据解读中,SpliceAI 的应用主要集中在以下几个方面:
1. 剪接变异的致病性预测
-
功能评估:SpliceAI 可以预测基因变异(如单核苷酸变异 SNVs、插入缺失 indels)是否会影响剪接位点或分支点的识别,从而改变 mRNA 的剪接模式,有助于判断变异的致病性。
-
优先级排序:在临床外显子组或基因组测序中,SpliceAI 可帮助从大量变异中筛选出可能影响剪接的候选致病变异,辅助遗传诊断。
2. 解读意义未明的变异(VUS)
-
许多临床检测中发现的变异被归类为“意义未明”(VUS)。SpliceAI 可以通过预测这些变异对剪接的影响(如激活隐秘剪接位点或破坏正常剪接位点),为 VUS 的重新分类提供证据。
3. 非编码变异的分析
-
传统上,非编码区域的变异(如内含子或深内含子区域)难以解读。SpliceAI 能够预测这些区域中可能影响剪接的变异(如深内含子变异导致新的外显子插入),扩展了临床变异的检测范围。
4. 辅助遗传病诊断
-
对于疑似遗传病患者,SpliceAI 可与其他工具(如 CADD、REVEL)结合,提高剪接相关致病变异的检出率。
5. 研究新发剪接位点
-
某些疾病可能由新发剪接事件(如外显子跳跃、内含子保留)驱动。SpliceAI 可模拟变异后的剪接模式,帮助发现新的致病机制。
6. 局限性及注意事项
-
假阳性/假阴性:SpliceAI 的预测仅仅是计算层面的预测,需要实验验证(如RNA测序或 minigene)。
-
组织特异性:剪接调控具有组织特异性,但 SpliceAI 的训练数据可能未覆盖所有组织。
-
结合其他证据:需遵循 ACMG/AMP 指南,综合多种证据(如种群频率、保守性、表型匹配)进行变异解读。
总之,SpliceAI 通过量化变异对剪接的影响(以 Δ score 表示),为临床遗传诊断提供了重要的功能预测证据,尤其对非编码变异的解读具有独特价值,最终结果需谨慎结合其他生化和临床数据。

DAMO开发者矩阵,由阿里巴巴达摩院和中国互联网协会联合发起,致力于探讨最前沿的技术趋势与应用成果,搭建高质量的交流与分享平台,推动技术创新与产业应用链接,围绕“人工智能与新型计算”构建开放共享的开发者生态。
更多推荐
 已为社区贡献7条内容
已为社区贡献7条内容





所有评论(0)