UltraLight-VM-UNet训练自建数据集
UltraLight-VM-UNet是一种轻量级医学图像分割网络,融合UNet架构与Mamba模型,参数量仅0.049M。该模型采用并行视觉Mamba层降低计算负载,结合注意力桥接模块提升分割性能。安装需配置Python≥3.8、PyTorch≥1.13及Mamba-SSM依赖。训练过程包括数据集准备、生成训练集、超参数设置等步骤,支持使用预训练权重。测试表明该模型在皮肤病数据集上表现优异,适用于
UltraLight-VM-UNet训练自建数据集
一、UltraLight-VM-UNet介绍
1.1模型简介
UltraLight-VM-UNet是一种在2024年提出的图像分割网络,主要用于医学影像分割,它采用了UNet的经典编解码结构,在编码器解码器上融合了Mamba这一新型架构,参数量仅0.049M,浮点运算数FLOPs为0.06G。
在皮肤病数据集ISIC2017、ISIC2018与PH2上,与其他模型比较性能指标如下: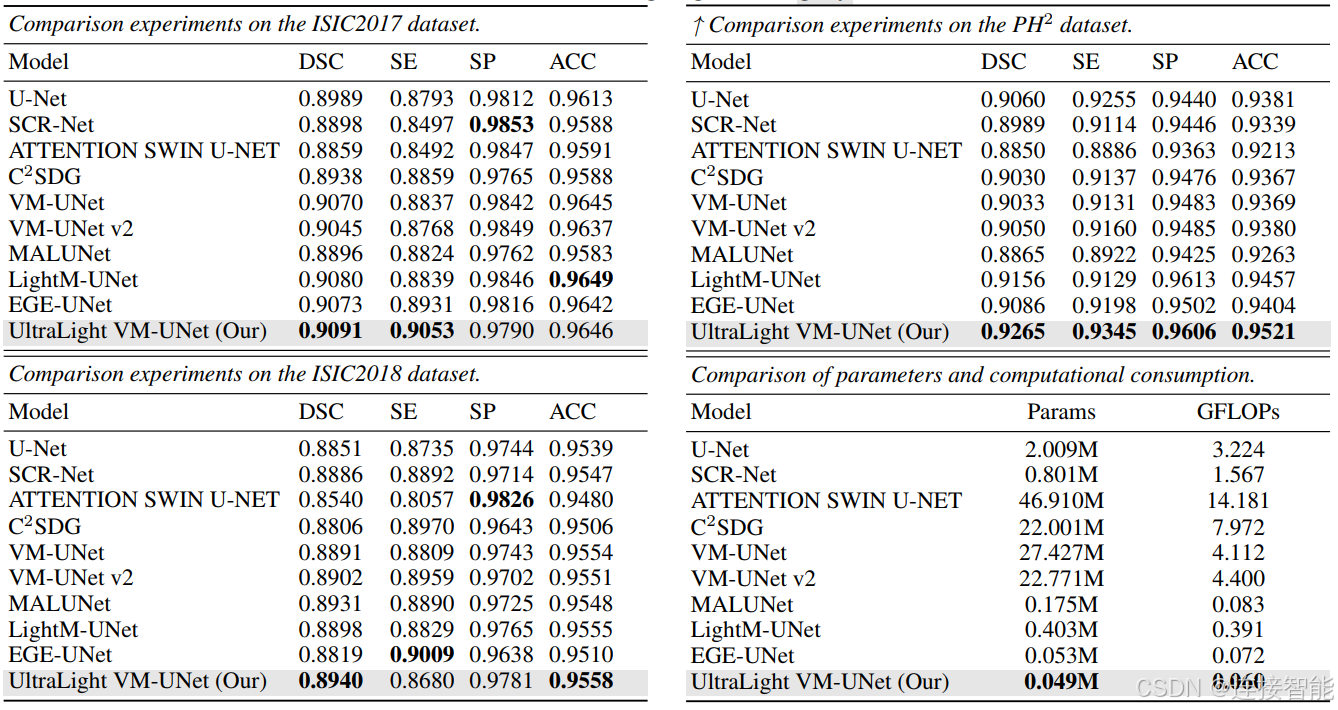
相关论文地址:https://arxiv.org/abs/2403.20035
Github项目地址:https://github.com/wurenkai/UltraLight-VM-UNet
1.2结构简介
模型整体结构如下: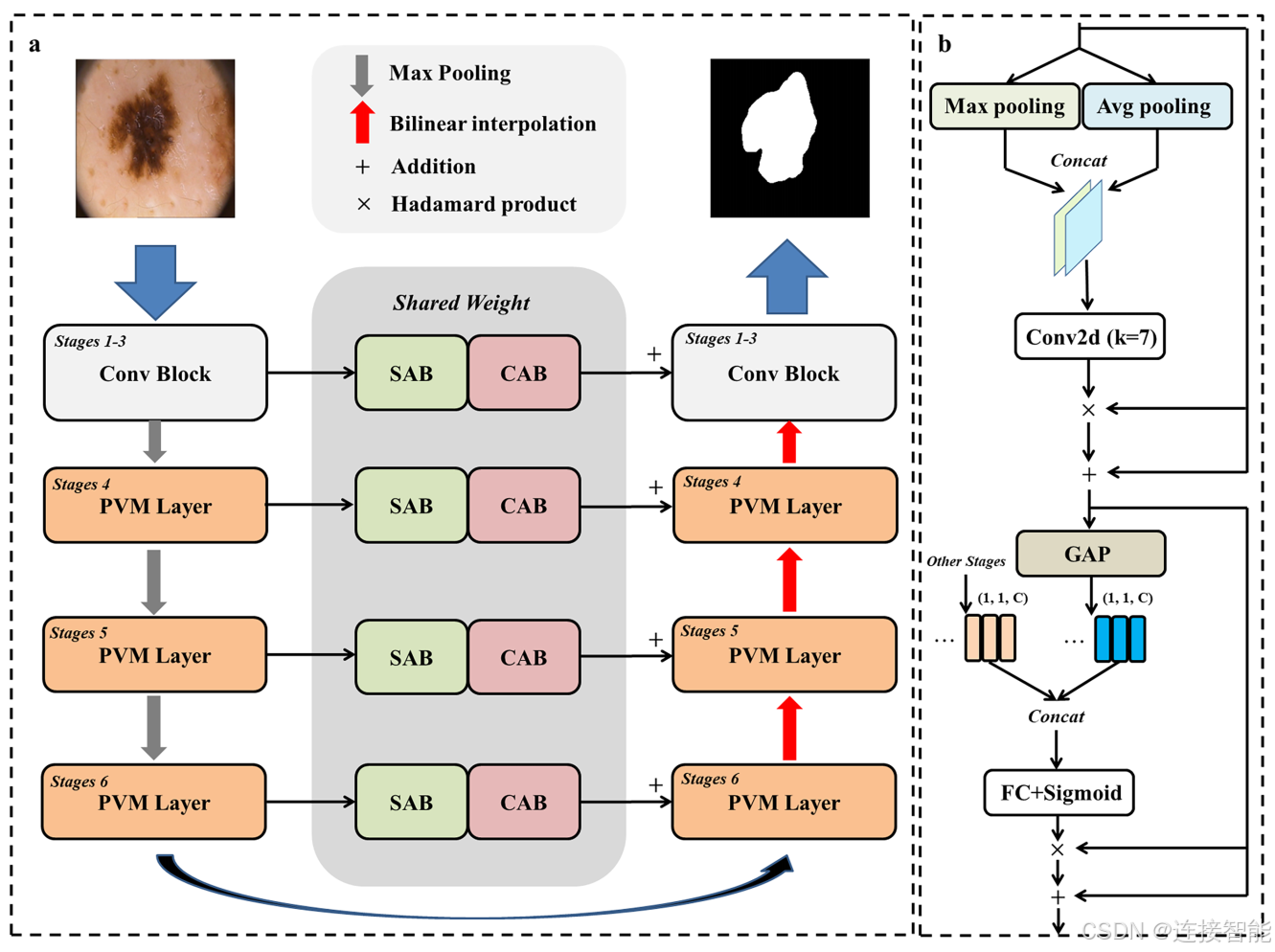
1.2.1采用并行视觉Mamba层(PVM Layer)
PVM层,通过将输入特征分割成多个并行分支(通常为四个),每个分支处理的通道数为原始通道数的四分之一,从而显著降低了计算负载和参数数量。
每个分支通过VSS Block(Vision Mamba Block)进行处理,然后通过残差连接和调整因子进行优化,最后通过拼接操作合并回原始通道数的特征。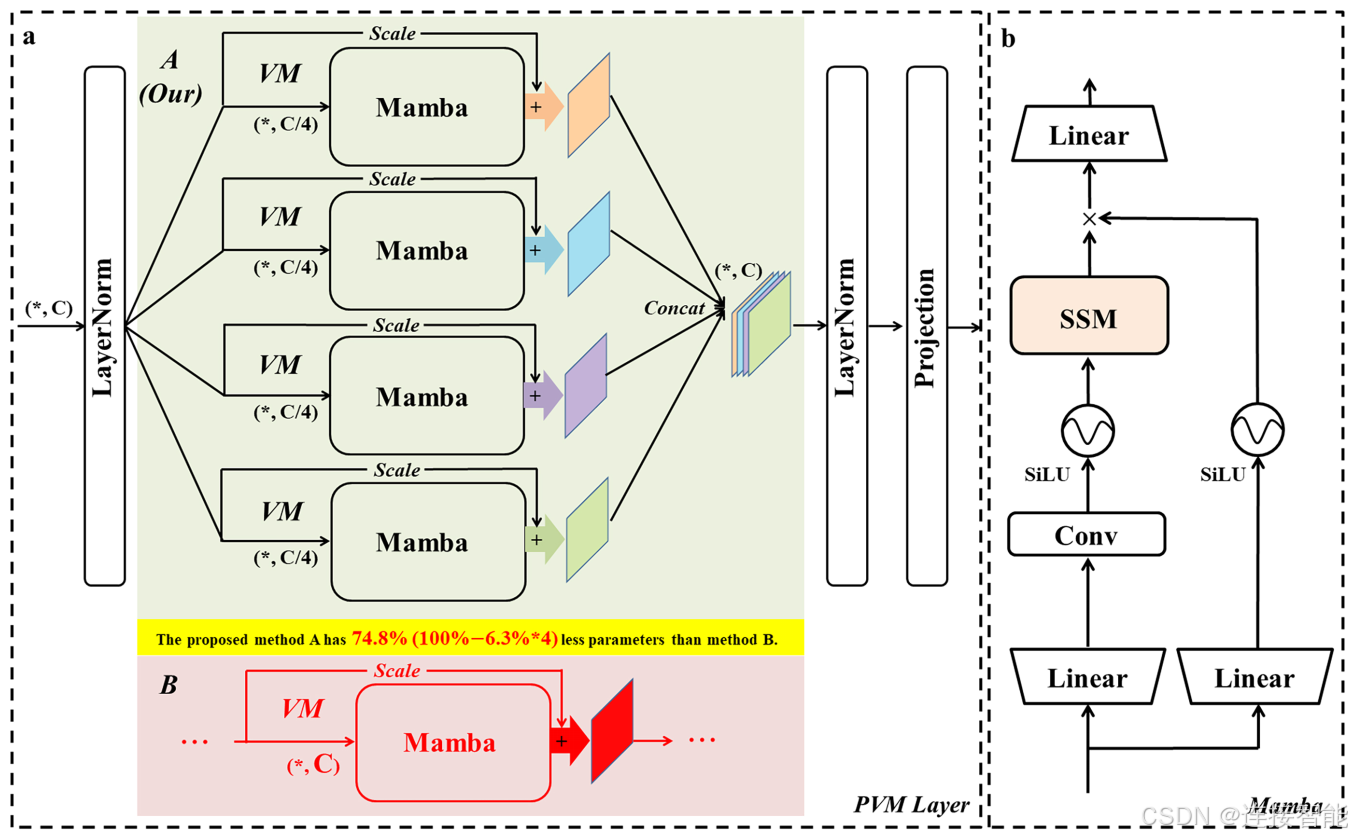
1.2.2融合UNet模型架构
采用了经典的U-Net架构,该架构包含一个编码器用于提取图像特征,和一个解码器用于根据提取的特征重建分割图。模型的前三层使用传统的卷积模块来提取浅层特征,而后三层则采用PVM来提取更深层次的特征。
1.2.3注意力桥接模块
在跳跃连接路径中,使用了通道注意力桥接(CAB)和空间注意力桥接(SAB)模块,以实现多尺度信息的融合。CAB模块通过全局平均池化、拼接操作、全连接层和Sigmoid激活函数来增强通道间的信息交互;SAB模块则包含最大池化、平均池化和扩展卷积等操作,以捕获空间上的重要特征。
注意力桥接模块的使用进一步提高了模型的收敛能力和对病变的敏感性,有助于提升分割性能。
二、UltraLight-VM-UNet安装
2.1环境与依赖包要求
根据Github上介绍,python版本应不低于3.8,pytorch版本不低于1.13,mamba_ssm版本不低于1.0.1。
本文安装环境如下:
系统:ubuntu22.04
pytorch:2.3.0+cu121
mamba-ssm:2.2.4
2.2安装过程
UltraLight-VM-UNet主要依赖pytorch包、mamba_ssm包,mamba安装较为困难,读者可参照本博客另一篇文章安装mamba:Mamba-ssm软件包安装。
主要依赖包安装结束后,使用Git命令下载UltraLight-VM-UNet项目源码,命令如下:
git clone https://github.com/wurenkai/UltraLight-VM-UNet.git
至此安装完成,UltraLight-VM-UNet安装难点主要在于mamba安装。
三、训练代码
3.1数据集准备
自建数据集应包含两个文件夹,一个为原始图片文件夹,一个为mask图片文件夹。原始图片应为3通道,mask为单通道,0灰度值表示背景,255灰度值表示目标。经实际测试1灰度值表示目标的mask图片也可正常训练,不必转换为255灰度值。
3.2生成训练集
进入项目源码文件夹下的dataprepare文件夹,打开Prepare_your_dataset.py,修改图片尺寸为实际图片尺寸,并且修改训练、验证、测试的图片数量,如下图:
修改完成后保存,并执行该文件,执行完毕后会在当前目录下生成6个npy后缀的文件,就是生成的训练集、验证集、测试集。
3.3超参数设置
进入项目源码文件夹下的configs文件夹,打开config_setting.py,修改图片尺寸、num_classes、batch_size、epochs等参数,如下:
3.4训练与测试
官方提供了在皮肤病数据集上训练的预训练权重文件,读者可以选择是否使用,下载地址为:https://drive.google.com/file/d/1lbyWRlDGCl5YL65V2-OwwZui2dHH8pH7/view
该预训练文件如何使用官网并未介绍,读者可自行摸索。
执行项目源码文件夹下的train.py文件开启训练,
python train.py
模型测试是执行文件夹下的test.py文件,只是训练得到的模型路径需要进行修改。

DAMO开发者矩阵,由阿里巴巴达摩院和中国互联网协会联合发起,致力于探讨最前沿的技术趋势与应用成果,搭建高质量的交流与分享平台,推动技术创新与产业应用链接,围绕“人工智能与新型计算”构建开放共享的开发者生态。
更多推荐
 已为社区贡献3条内容
已为社区贡献3条内容





所有评论(0)