UNET 细胞图像分割实战:从原理到代码实现
通过本文的实践,我们展示了如何使用 UNET 模型实现高精度的细胞图像分割。从数据准备、模型构建到训练和评估,每个步骤都对最终结果有着重要影响。未来,随着深度学习技术的不断发展,我们可以期待更强大的模型架构和更高效的训练方法,进一步推动生物医学图像分析领域的发展。结合 Transformer 架构提升长距离依赖建模能力利用半监督学习和弱监督学习处理标注数据不足的问题开发实时细胞分割系统,支持临床诊
一、生物医学图像分割的重要性
在现代生物医学研究中,细胞图像分割是一项关键技术。它能够帮助医生和研究人员:
- 精确测量细胞尺寸、形态和数量
- 识别病变细胞,辅助疾病诊断(如癌症筛查)
- 追踪细胞分裂和迁移过程
- 分析药物对细胞的影响
传统的手动分割方法不仅耗时耗力,而且容易受到主观因素影响。而基于深度学习的 UNET 模型,为细胞图像分割提供了高精度、自动化的解决方案。
二、UNET 模型架构详解
2.1 核心结构:编码器 - 解码器
UNET 由一个编码器路径(收缩路径)和一个解码器路径(扩展路径)组成,形似英文字母 "U":
- 编码器:通过卷积和池化操作逐步减少空间维度(下采样),提取图像特征
- 解码器:通过反卷积和跳跃连接逐步恢复空间维度(上采样),精确定位目标区域
- 跳跃连接:将编码器的特征图直接连接到解码器对应层级,保留细节信息
这种设计使得 UNET 能够同时捕获全局上下文信息和局部细节特征,特别适合生物医学图像分割任务。
2.2 对比传统分割方法的优势
与传统方法(如阈值分割、边缘检测)相比,UNET 的优势体现在:
- 端到端训练,无需手动设计特征
- 能够处理复杂的细胞形态和重叠情况
- 对噪声和光照变化具有更强的鲁棒性
- 可扩展性强,适用于不同类型的生物医学图像
三、细胞图像分割实战
3.1 数据集准备
我们使用BBBC005细胞图像数据集,包含 30 张 HeLa 细胞荧光显微图像及对应的分割掩码:
from torch.utils.data import Dataset
import PIL.Image as Image
import os
import numpy as np
import torch
from torch import nn
from torch.utils.data import DataLoader
from torch import nn, optim
from torchvision.transforms import transforms
from tqdm import tqdm
import skimage.io as io3.2 模型实现
使用 PyTorch 实现 UNET 模型:
def train_dataset(img_root, label_root):
imgs = []
n = os.listdir(img_root)
for i in n:
img = os.path.join(img_root, i)
label = os.path.join(label_root, i)
imgs.append((img, label))
return imgs
class TrainDataset(Dataset):
def __init__(self, img_root, label_root, transform=None, target_transform=None):
imgs = train_dataset(img_root, label_root)
self.imgs = imgs
self.transform = transform
self.target_transform = target_transform
def __getitem__(self, index):
x_path, y_path = self.imgs[index]
img_x = Image.open(x_path)
img_y = Image.open(y_path)
if self.transform is not None:
img_x = self.transform(img_x)
if self.target_transform is not None:
img_y = self.target_transform(img_y)
return img_x, img_y
def __len__(self):
return len(self.imgs)
def test_dataset(img_root, label_root):
imgs = []
n = os.listdir(img_root) # os.listdir 获取 img_root 目录下的所有文件列表
for i in n:
img = os.path.join(img_root, i) # 通过 os.path.join 构建图像文件的完整路径 img_root/0.png
label = os.path.join(label_root, i) # 通过 os.path.join 构建标签文件的完整路径 label_root/0_mask.png
imgs.append((img, label)) # 将图像路径和标签路径作为一个元组添加到 imgs 列表
return imgs
class TestDataset(Dataset):
def __init__(self, img_root, label_root, transform=None, target_transform=None):
imgs = test_dataset(img_root, label_root)
self.imgs = imgs
self.transform = transform
self.target_transform = target_transform
def __getitem__(self, index):
x_path, y_path = self.imgs[index]
img_x = Image.open(x_path)
img_y = Image.open(y_path)
if self.transform is not None:
img_x = self.transform(img_x)
if self.target_transform is not None:
img_y = self.target_transform(img_y)
return img_x, img_y
def __len__(self):
return len(self.imgs)3.3 训练过程
Sky = [128,128,128]
Building = [128,0,0]
Pole = [192,192,128]
Road = [128,64,128]
Pavement = [60,40,222]
Tree = [128,128,0]
SignSymbol = [192,128,128]
Fence = [64,64,128]
Car = [64,0,128]
Pedestrian = [64,64,0]
Bicyclist = [0,128,192]
Unlabelled = [0,0,0]
COLOR_DICT = np.array([Sky, Building, Pole, Road, Pavement,
Tree, SignSymbol, Fence, Car, Pedestrian, Bicyclist, Unlabelled])
#将单通道的分类标签图像(每个像素值为类别索引) 转换为彩色图像, 颜色由 color_dict 定义。
def labelVisualize(num_class,color_dict,img):
img = img[:,:,0] if len(img.shape) == 3 else img
img_out = np.zeros(img.shape + (3,))
for i in range(num_class):
img_out[img == i,:] = color_dict[i]
return img_out / 255
class DoubleConv(nn.Module):
def __init__(self, in_ch, out_ch):
super(DoubleConv, self).__init__()
self.conv = nn.Sequential(
nn.Conv2d(in_ch, out_ch, 3, padding=1),
nn.BatchNorm2d(out_ch),
nn.ReLU(inplace=True),
nn.Conv2d(out_ch, out_ch, 3, padding=1),
nn.BatchNorm2d(out_ch),
nn.ReLU(inplace=True)
)
def forward(self, input):
return self.conv(input)
class Unet(nn.Module):
def __init__(self, in_ch, out_ch):
super(Unet, self).__init__()
self.conv1 = DoubleConv(in_ch, 64)
self.pool1 = nn.MaxPool2d(2)
self.conv2 = DoubleConv(64, 128)
self.pool2 = nn.MaxPool2d(2)
self.conv3 = DoubleConv(128, 256)
self.pool3 = nn.MaxPool2d(2)
self.conv4 = DoubleConv(256, 512)
self.pool4 = nn.MaxPool2d(2)
self.conv5 = DoubleConv(512, 1024)
self.up6 = nn.ConvTranspose2d(1024, 512, 2, stride=2)
self.conv6 = DoubleConv(1024, 512)
self.up7 = nn.ConvTranspose2d(512, 256, 2, stride=2)
self.conv7 = DoubleConv(512, 256)
self.up8 = nn.ConvTranspose2d(256, 128, 2, stride=2)
self.conv8 = DoubleConv(256, 128)
self.up9 = nn.ConvTranspose2d(128, 64, 2, stride=2)
self.conv9 = DoubleConv(128, 64)
self.conv10 = nn.Conv2d(64, out_ch, 1)
def forward(self, x):
c1 = self.conv1(x)
p1 = self.pool1(c1)
c2 = self.conv2(p1)
p2 = self.pool2(c2)
c3 = self.conv3(p2)
p3 = self.pool3(c3)
c4 = self.conv4(p3)
p4 = self.pool4(c4)
c5 = self.conv5(p4)
up_6 = self.up6(c5)
merge6 = torch.cat([up_6, c4], dim=1)
c6 = self.conv6(merge6)
up_7 = self.up7(c6)
merge7 = torch.cat([up_7, c3], dim=1)
c7 = self.conv7(merge7)
up_8 = self.up8(c7)
merge8 = torch.cat([up_8, c2], dim=1)
c8 = self.conv8(merge8)
up_9 = self.up9(c8)
merge9 = torch.cat([up_9, c1], dim=1)
c9 = self.conv9(merge9)
c10 = self.conv10(c9)
return c10
# 是否使用cuda
device = torch.device("cuda" if torch.cuda.is_available() else "cpu")
x_transforms = transforms.Compose([
transforms.ToTensor(),
transforms.Normalize([0.5], [0.5])
])
# mask只需要转换成tensor
mask_transforms = transforms.ToTensor()
model = Unet(1, 1).to(device)
batch_size = 4
criterion = nn.BCEWithLogitsLoss() # BCELoss
optimizer = optim.Adam(model.parameters(), lr=0.001)
imgs, labels = next(iter(train_dataloader))
imgs.shape
四、结果可视化与评估
4.1 分割结果可视化
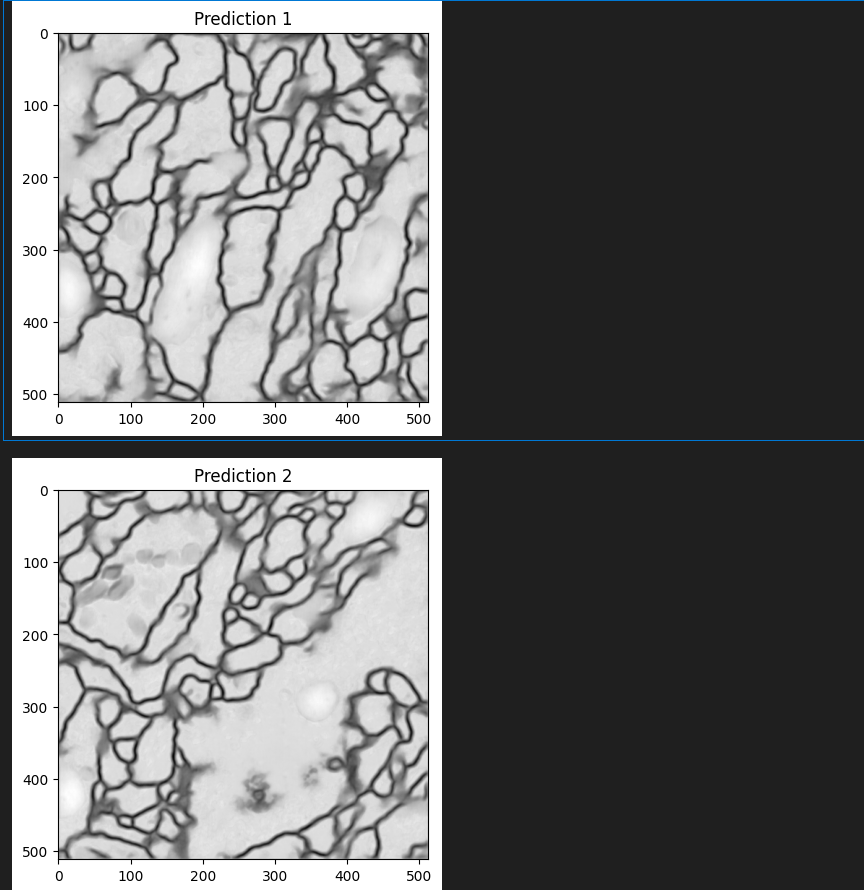
五、应用拓展与优化方向
5.1 多类别细胞分割
通过修改输出通道数和使用 softmax 激活函数,可以扩展 UNET 用于多类别细胞分割:
# 修改模型定义,设置n_classes为细胞类型数量
model = UNet(n_channels=1, n_classes=3) # 假设3种细胞类型
criterion = nn.CrossEntropyLoss()5.2 数据增强
使用 albumentations 库增强训练数据:
import albumentations as A
transform = A.Compose([
A.HorizontalFlip(p=0.5),
A.VerticalFlip(p=0.5),
A.Rotate(limit=45, p=0.5),
A.RandomBrightnessContrast(p=0.2),
])
# 在数据加载时应用增强
def load_data_with_augmentation(data_dir):
# ... 其余代码保持不变
for img_name in os.listdir(os.path.join(data_dir, 'images')):
# ... 加载图像和掩码
augmented = transform(image=np.array(img), mask=np.array(mask))
img = augmented['image']
mask = augmented['mask']
# ... 继续处理5.3 预训练模型与迁移学习
使用在大型医学图像数据集上预训练的 UNET 模型,可以显著提高分割性能:
# 使用segmentation_models_pytorch库加载预训练模型
import segmentation_models_pytorch as smp
model = smp.Unet(
encoder_name="resnet34", # 使用ResNet34作为编码器
encoder_weights="imagenet", # 使用ImageNet预训练权重
in_channels=1, # 输入通道数
classes=1, # 输出类别数
)六、总结与展望
通过本文的实践,我们展示了如何使用 UNET 模型实现高精度的细胞图像分割。从数据准备、模型构建到训练和评估,每个步骤都对最终结果有着重要影响。
未来,随着深度学习技术的不断发展,我们可以期待更强大的模型架构和更高效的训练方法,进一步推动生物医学图像分析领域的发展。例如:
- 结合 Transformer 架构提升长距离依赖建模能力
- 利用半监督学习和弱监督学习处理标注数据不足的问题
- 开发实时细胞分割系统,支持临床诊断决策
通过不断探索和创新,我们相信基于 UNET 的图像分割技术将在生物医学研究和临床应用中发挥越来越重要的作用。
七、常见挑战与解决方案
7.1 细胞重叠问题
在实际细胞图像中,细胞常常会出现重叠现象,这给分割带来了很大挑战。解决方法包括:
八、前沿研究进展
8.1 Transformer 与 UNET 的结合
最近的研究表明,将 Transformer 架构引入 UNET 可以显著提升分割性能。例如:
8.2 医学图像分割的自监督学习
由于医学图像标注成本高,自监督学习方法受到越来越多关注:
8.3 少样本学习在细胞分割中的应用
当标注数据有限时,可以使用少样本学习技术:
九、实际应用案例
9.1 癌细胞检测与分析
某癌症研究中心使用 UNET 模型对病理切片中的癌细胞进行分割,辅助医生进行癌症分级和预后评估。系统能够自动计算癌细胞密度、核质比等重要指标,为临床决策提供数据支持。
9.2 药物研发中的细胞分析
制药公司在药物研发过程中,需要分析药物对细胞的影响。通过 UNET 分割技术,能够精确测量细胞形态变化、增殖速率和凋亡率,加速药物筛选过程。
9.3 神经科学中的神经元追踪
在神经科学研究中,需要对神经元进行三维重建和追踪。结合 UNET 和 3D 卷积,研究人员能够自动分割显微镜图像中的神经元,大大提高了分析效率。
十、资源推荐
10.1 数据集
10.2 开源工具
10.3 学术论文
十一、常见问题解答
分享
- 实例分割技术:使用 Mask R-CNN 等模型同时检测和分割每个细胞实例
- 边缘增强:在损失函数中加入边缘检测分支,强化细胞边界特征
- 距离变换:将分割问题转化为距离场回归问题,通过分水岭算法分离重叠细胞
# 距离变换示例代码 from scipy import ndimage def watershed_segmenation(mask): # 计算距离变换 distance = ndimage.distance_transform_edt(mask) # 寻找局部最大值作为种子点 local_maxi = peak_local_max(distance, indices=False, footprint=np.ones((3, 3)), labels=mask) markers = ndimage.label(local_maxi)[0] # 应用分水岭算法 labels = watershed(-distance, markers, mask=mask) return labels7.2 数据不平衡问题
在某些细胞分割任务中,目标细胞可能只占图像的很小一部分,导致类别不平衡。解决策略有:
- 加权损失函数:对小目标区域赋予更高的权重
- 数据重采样:通过过采样少数类或欠采样多数类来平衡数据分布
- 焦点损失 (Focal Loss):降低简单样本的权重,专注于难分类样本
# 焦点损失实现 class FocalLoss(nn.Module): def __init__(self, alpha=0.25, gamma=2): super(FocalLoss, self).__init__() self.alpha = alpha self.gamma = gamma def forward(self, inputs, targets): BCE_loss = F.binary_cross_entropy_with_logits(inputs, targets, reduction='none') pt = torch.exp(-BCE_loss) F_loss = self.alpha * (1-pt)**self.gamma * BCE_loss return F_loss.mean()7.3 小目标细胞检测
对于尺寸特别小的细胞或细胞器,分割难度较大。可以采用以下方法:
- 多尺度特征融合:结合浅层高分辨率特征和深层语义特征
- 注意力机制:引导模型关注小目标区域
- 图像金字塔:在不同尺度下处理图像,增强小目标检测能力
- UNETR:使用 Vision Transformer 作为编码器,能够捕获长距离依赖关系
- Swin-UNET:采用 Shifted Windows Transformer,提高了局部特征表示能力
- 对比学习:通过图像旋转、裁剪等变换构建正负样本对
- 掩码自编码器 (MAE):通过重构被掩码的图像区域学习特征表示
- ** pretext 任务 **:设计诸如拼图、颜色预测等辅助任务来学习图像特征
- 元学习:通过在多个相关任务上训练,快速适应新的分割任务
- 原型网络:学习类别原型,根据与原型的相似度进行分割
- 条件生成对抗网络:生成合成数据以扩充训练集
- BBBC005:HeLa 细胞荧光显微图像数据集
- Cellpose Dataset:包含多种细胞类型的分割数据集
- TNBC_Nuclei:乳腺癌细胞核分割数据集
- Cellpose:基于深度学习的细胞分割工具
- Stardist:使用星形多边形表示的细胞分割方法
- Segmentation Models Pytorch:包含多种预训练分割模型的库
- Ronneberger et al., "U-Net: Convolutional Networks for Biomedical Image Segmentation", 2015
- Chen et al., "DeepLabv3+: Encoder-Decoder with Atrous Separable Convolution for Semantic Segmentation", 2018
- Hatamizadeh et al., "UNETR: Transformers for 3D Medical Image Segmentation", 2021
-
问:UNET 适合处理大尺寸图像吗?
答:直接处理大尺寸图像会导致内存不足。可以采用滑动窗口策略或图像金字塔方法。 -
问:如何提高 UNET 的分割精度?
答:可以尝试使用更深的网络架构、更多的数据增强、预训练模型或结合注意力机制。 -
问:训练 UNET 需要多少数据?
答:这取决于问题的复杂度。通常建议至少有几百张标注图像,如果数据有限,可以考虑迁移学习或数据合成技术。 -
问:如何处理医学图像中的噪声?
答:可以在数据预处理阶段应用滤波方法,或在网络中加入噪声鲁棒性训练。 -
问:UNET 可以用于视频中的细胞追踪吗?
答:可以,结合光流估计或时序信息,UNET 可以扩展到视频序列的细胞分割和追踪。

DAMO开发者矩阵,由阿里巴巴达摩院和中国互联网协会联合发起,致力于探讨最前沿的技术趋势与应用成果,搭建高质量的交流与分享平台,推动技术创新与产业应用链接,围绕“人工智能与新型计算”构建开放共享的开发者生态。
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容








所有评论(0)