医学图像笔记(三)NIFTI数据格式
医学图像笔记(二)NIFTI数据格式1、NIFTI格式的基本内容2、python读取.nii.gz文件1、NIFTI格式的基本内容“神经成像信息技术创新”将NIFTI格式视为ANALYZE7.5格式的替代品。NIFTI最初是用于神经成像的,但它也适用于一些其他的领域。NIFTI中一个主要的特点在于它包含了两个仿射坐标定义,这两个仿射坐标定义能够将每个立体元素指标(i,j,k)和空间位置(x,y,z
医学图像笔记(三)NIFTI数据格式
1、NIFTI格式的基本内容
“神经成像信息技术创新”将NIFTI格式视为ANALYZE7.5格式的替代品。NIFTI最初是用于神经成像的,但它也适用于一些其他的领域。NIFTI中一个主要的特点在于它包含了两个仿射坐标定义,这两个仿射坐标定义能够将每个立体元素指标(i,j,k)和空间位置(x,y,z)联系起来。 Nibabel是用于读取nifti文件的一个朋友Python库,“oro.nifti”是用于读取nifti数据的一个R工具包。
标准NIfTI图像的扩展名是.nii,包含了头文件及图像资料。由于NIfTI格式和Analyze格式的关系,因此NIfTI格式也可使用独立的图像文件[.img]和头文件[.hdr]。单独的.nii格式文件的优势就是可以用标准的压缩软件[如gzip],而且一些分析软件包[比如FSL]可以直接读取和写入压缩的.nii文件[扩展名为.nii.gz]。
简而言之,nii格式和.nii.gz格式是一个东西。
2、python使用 nibabel读取.nii.gz文件
# -*- coding : UTF-8 -*-
# @file : rd_niigz.py
# @Time : 2021-09-14 17:40
# @Author : wmz
# encoding=utf8
'''
查看和显示nii文件
'''
import matplotlib
matplotlib.use('TkAgg')
from matplotlib import pylab as plt
import nibabel as nib
from nibabel import nifti1
from nibabel.viewers import OrthoSlicer3D
# 文件名,nii或nii.gz
example_filename = './img/0001.nii.gz'
img = nib.load(example_filename)
print(img)
print(img.header['db_name']) # 输出头信息
# shape不一定只有三个参数,打印出来看一下
width, height, queue = img.dataobj.shape
# 显示3D图像
OrthoSlicer3D(img.dataobj).show()
num = 1
# 按照10的步长,切片,显示2D图像
for i in range(0, queue, 10):
img_arr = img.dataobj[:, :, i]
plt.subplot(5, 4, num)
plt.imshow(img_arr, cmap='gray')
num += 1
plt.show()
对于图像处理,可用信息包括:
| data shape | (400, 256, 256) |
|---|---|
| affine | [[ 1. 0. 0. -213.39100647] [ 0. 1. 0. 47.1853981 ] [ 0. 0. 1. -561.7199707 ] [ 0. 0. 0. 1. ]] |
| datatype | int16 |
| xyzt_units | 2 |
| qoffset_x | -213.391 |
| qoffset_y | 47.1854 |
| qoffset_z | -561.72 |
打印输出:
D:\anaconda3\envs\pytorch190\python.exe D:/documents/python/md_img/rd_niigz.py
<class 'nibabel.nifti1.Nifti1Image'>
data shape (400, 256, 256)
affine:
[[ 1. 0. 0. -213.39100647]
[ 0. 1. 0. 47.1853981 ]
[ 0. 0. 1. -561.7199707 ]
[ 0. 0. 0. 1. ]]
metadata:
<class 'nibabel.nifti1.Nifti1Header'> object, endian='<'
sizeof_hdr : 348
data_type : b''
db_name : b''
extents : 0
session_error : 0
regular : b'r'
dim_info : 0
dim : [ 3 400 256 256 1 1 1 1]
intent_p1 : 0.0
intent_p2 : 0.0
intent_p3 : 0.0
intent_code : none
datatype : int16
bitpix : 16
slice_start : 0
pixdim : [1. 1. 1. 1. 0. 0. 0. 0.]
vox_offset : 0.0
scl_slope : nan
scl_inter : nan
slice_end : 0
slice_code : unknown
xyzt_units : 2
cal_max : 0.0
cal_min : 0.0
slice_duration : 0.0
toffset : 0.0
glmax : 0
glmin : 0
descrip : b''
aux_file : b''
qform_code : scanner
sform_code : unknown
quatern_b : 0.0
quatern_c : 0.0
quatern_d : 0.0
qoffset_x : -213.391
qoffset_y : 47.1854
qoffset_z : -561.72
srow_x : [0. 0. 0. 0.]
srow_y : [0. 0. 0. 0.]
srow_z : [0. 0. 0. 0.]
intent_name : b''
magic : b'n+1'
b''
显示: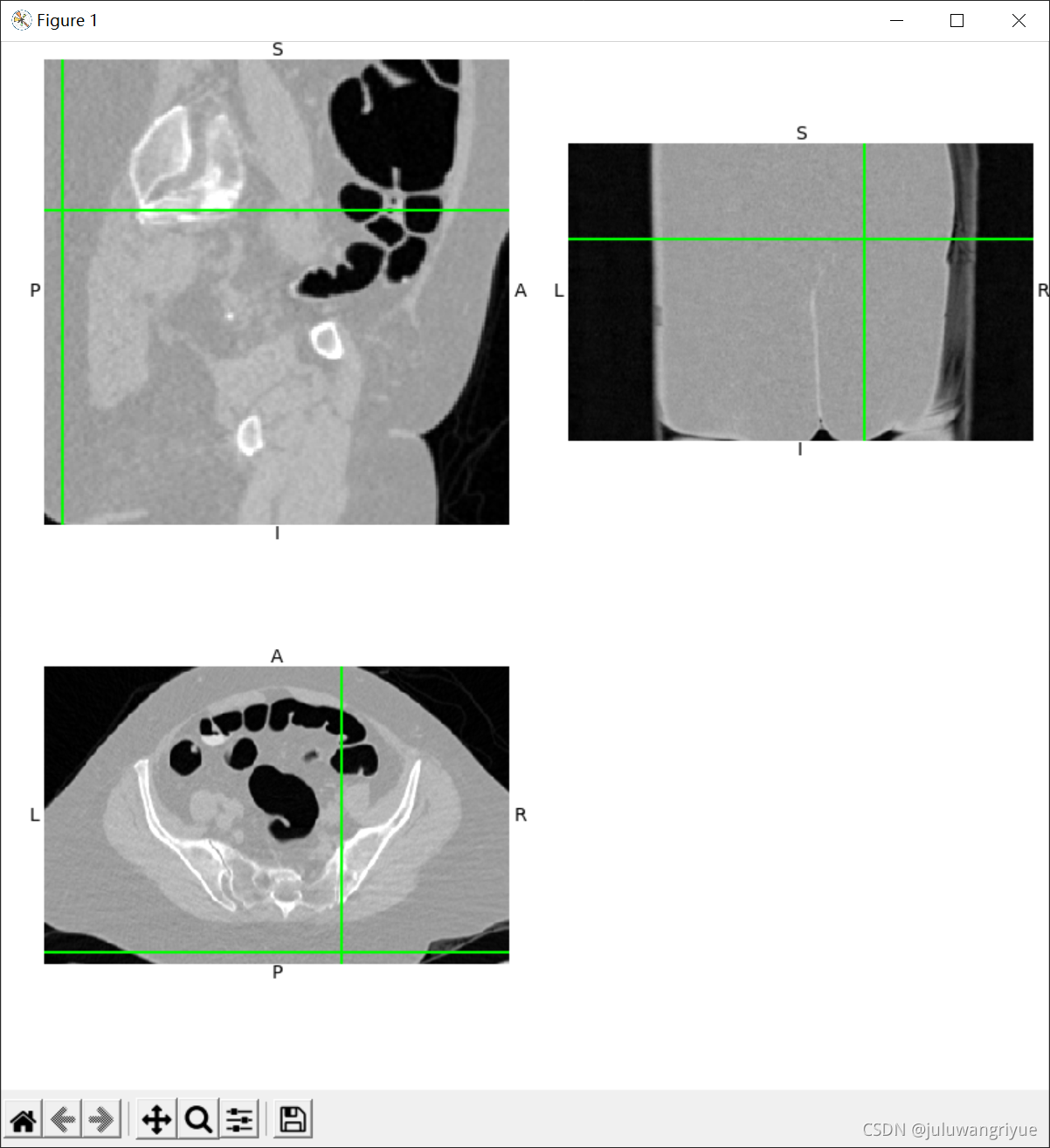
3、python使用itk读取.nii,gz文件
# -*- coding : UTF-8 -*-
# @file : itk_p3.py
# @Time : 2021-09-14 18:13
# @Author : wmz
import SimpleITK as sitk
import skimage.io as io
def read_img(path):
img = sitk.ReadImage(path)
data = sitk.GetArrayFromImage(img)
return data
# 显示一个系列图
def show_img(data):
for i in range(data.shape[0]):
io.imshow(data[i, :, :], cmap='gray')
print(i)
io.show()
# 单张显示
# def show_img(ori_img):
# io.imshow(ori_img[100], cmap='gray')
# io.show()
if __name__ == "__main__":
# window下的文件夹路径
path = '..\\md_img\\img\\0001.nii.gz'
data = read_img(path)
show_img(data)
显示: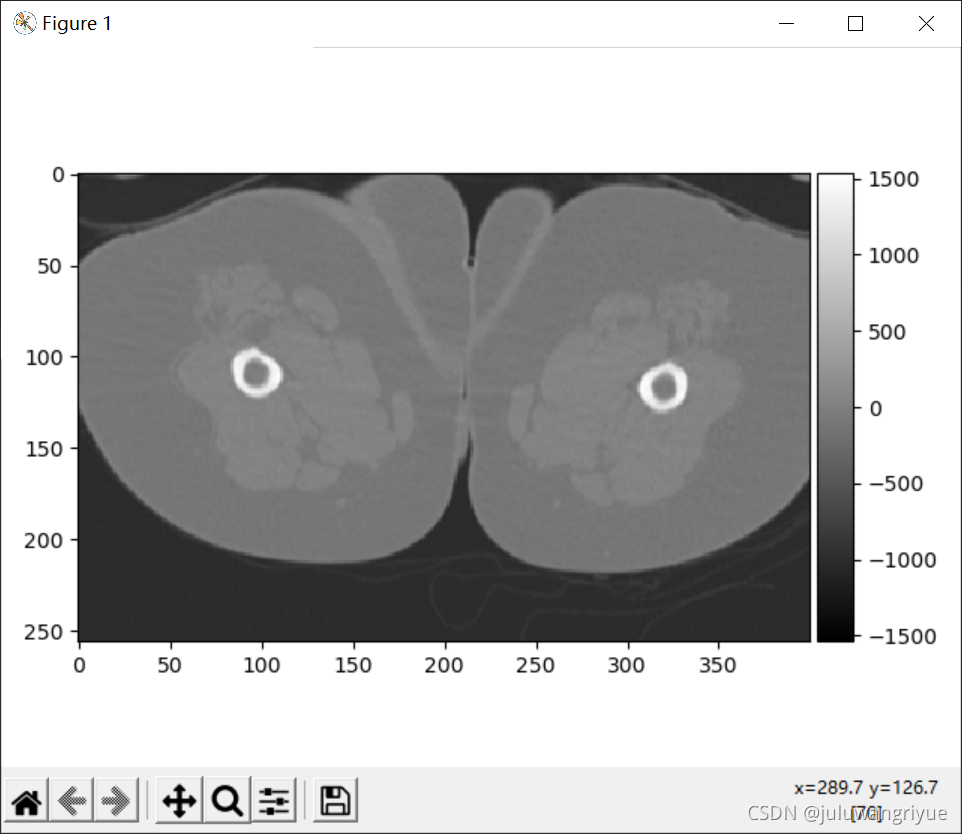
调试信息: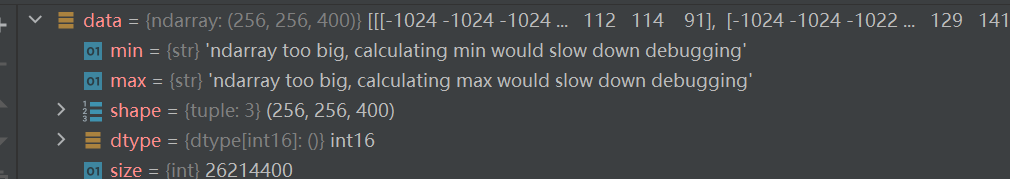
通过与nibable读取的信息对比可知,nibable维度信息 是 x,y,z轴顺序,itk读取的信息是z,x,y轴顺序。
itk下调试获取其他信息: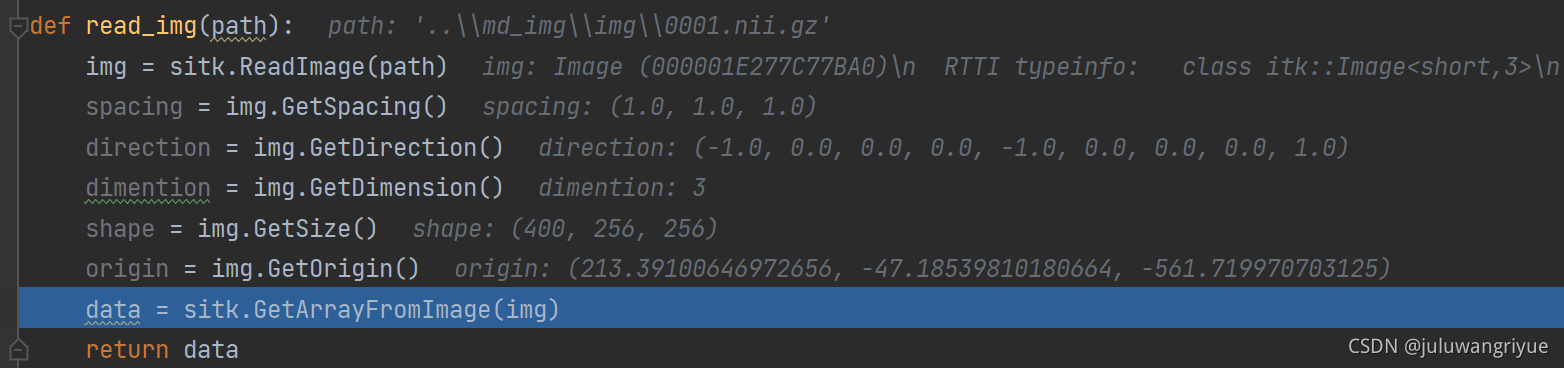

DAMO开发者矩阵,由阿里巴巴达摩院和中国互联网协会联合发起,致力于探讨最前沿的技术趋势与应用成果,搭建高质量的交流与分享平台,推动技术创新与产业应用链接,围绕“人工智能与新型计算”构建开放共享的开发者生态。
更多推荐
 已为社区贡献7条内容
已为社区贡献7条内容







所有评论(0)