TCGA数据库筛选逻辑
TCGA数据库筛选逻辑
目录
为什么第一个筛选范围是从Experimental Strategy这里开始,而不是大家所熟知的,更加清晰的数据类别 Data Type(数据类型)开始?
为什么从 Experimental Strategy(实验策略)开始?
第一次接触生信领域,TCGA数据库是个非常好的研究数据平台,最近为了准备ppt给老师汇报,要好好地了解一下TCGA的筛选逻辑策略,每一个筛选条目是什么,整理了一下,想了解的同学们可以了解一下看看有没有帮助!以下类别筛选是以选中肺部数据为前提。

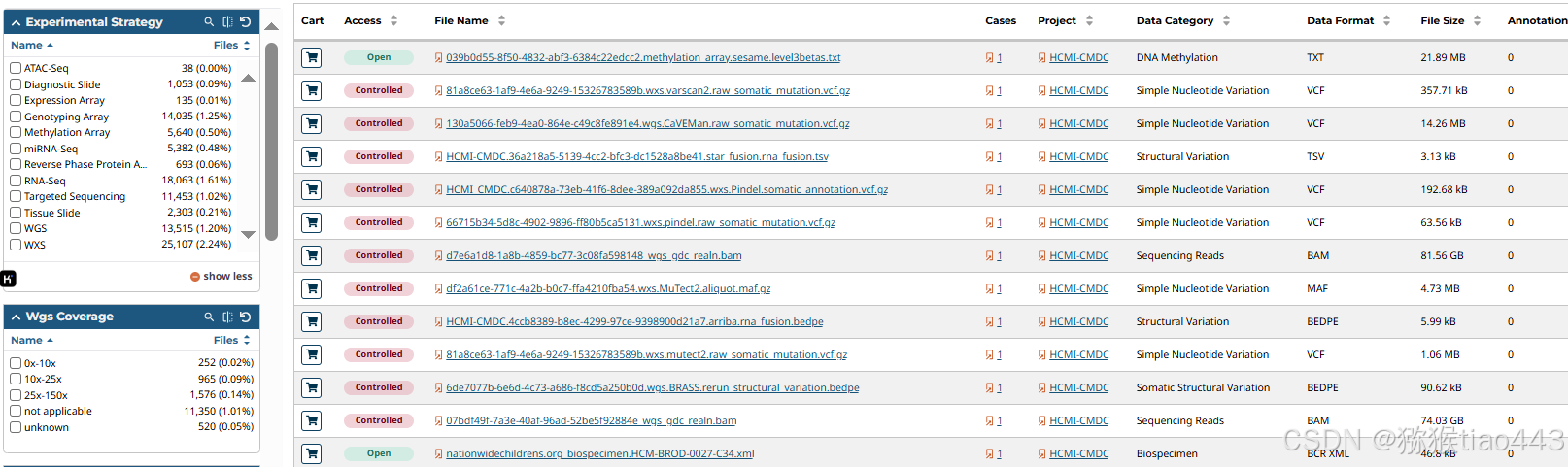
1.Experimental Strategy(实验策略)
- ATAC-Seq(Assay for Transposase-Accessible Chromatin using sequencing):用于检测染色质可及性,研究基因调控。
- Diagnostic Slide(诊断切片):组织学切片影像数据,可用于病理分析。
- Expression Array(表达芯片):基因表达分析技术。
- Genotyping Array(基因分型芯片):用于检测DNA变异,如SNPs。
- Methylation Array(甲基化芯片):检测DNA甲基化水平。
- miRNA-Seq(microRNA测序):检测miRNA的表达情况。
- Reverse Phase Protein Array(RPPA):基于抗体检测蛋白表达水平。
- RNA-Seq(RNA测序):用于研究转录组数据,分析基因表达情况。
- Targeted Sequencing(靶向测序):针对特定基因或基因组区域进行测序。
- Tissue Slide(组织切片):病理组织影像数据。
- WGS(Whole Genome Sequencing,全基因组测序):测序整个基因组。
- WXS(Whole Exome Sequencing,全外显子组测序):仅测序外显子区域(编码区)
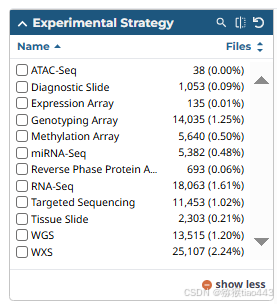
2. WGS Coverage(全基因组测序覆盖度)
用于WGS数据,表示测序深度(coverage),即一个碱基被测序的平均次数:
- 0x-10x:覆盖度较低,可能适用于群体研究。
- 10x-25x:中等覆盖度,适用于较大样本量的基因变异分析。
- 25x-150x:高覆盖度,更适合精确突变检测和个体层面的分析。
- not applicable:不适用于非WGS数据。
- unknown:未知覆盖度.
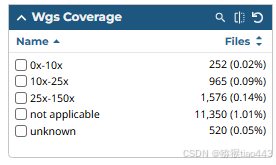
3. Data Category(数据类别)
表示数据的大类:
- Biospecimen(生物样本):关于组织样本的信息,如组织类型、采集方式等。
- Clinical(临床数据):患者的临床信息,如年龄、性别、病理分期等。
- Copy Number Variation(拷贝数变异):基因组中DNA片段的拷贝数增减情况。
- DNA Methylation(DNA甲基化):DNA甲基化修饰信息。
- Proteome Profiling(蛋白组学分析):蛋白表达信息。
- Sequencing Reads(测序读段):原始测序数据。
- Simple Nucleotide Variation(单核苷酸变异):SNPs或小Indels变异信息。
- Somatic Structural Variation(体细胞结构变异):体细胞中的大规模结构变异。
- Structural Variation(结构变异):包括体细胞和生殖细胞的结构变异。
- Transcriptome Profiling(转录组分析):RNA表达相关数据。
| 数据类别 | 主要研究方向 | 数据内容 | 对应的实验策略(Experimental Strategy) |
|---|
| Biospecimen(生物样本数据) | 样本信息,如组织来源、处理方式 | 组织采样信息、生物样本信息 | N/A(非测序数据) |
| Clinical(临床数据) | 患者基本信息、诊断信息、预后等 | 诊断数据、治疗方案、生存情况 | N/A(非测序数据) |
| Copy Number Variation(拷贝数变异) | DNA 片段的扩增或缺失 | CNV 片段、拷贝数值 | Genotyping Array, WGS, WXS |
| DNA Methylation(DNA 甲基化) | 表观遗传修饰,影响基因表达 | CpG 位点甲基化值 | Methylation Array |
| Proteome Profiling(蛋白质组数据) | 蛋白表达水平 | 蛋白表达定量数据 | Reverse Phase Protein Array |
| Sequencing Reads(测序原始数据) | 原始测序数据 | FASTQ/BAM 文件 | RNA-Seq, WGS, WXS |
| Simple Nucleotide Variation(单核苷酸变异) | 基因突变(SNP、Indel) | 突变数据(VCF) | WGS, WXS, Targeted Sequencing |
| Somatic Structural Variation(体细胞结构变异) | 染色体重排、大片段插入缺失 | 结构变异(SV)数据 | WGS |
| Structural Variation(结构变异) | 体细胞 + 生殖系结构变异 | 结构变异数据(VCF) | WGS |
| Transcriptome Profiling(转录组数据) | 基因表达、可变剪切、miRNA 表达 | 基因表达定量(FPKM, TPM) | RNA-Seq, miRNA-Seq |
4. Data Type(数据类型)
数据类型是更具体的数据分类,例如:
- Aggregated Somatic Mutation(聚合的体细胞突变数据):来自多个研究汇总的突变信息。
- Aligned Reads(比对后的测序数据):经过比对的BAM格式数据。
- Allele-specific Copy Number Segment(等位基因特异性拷贝数片段):拷贝数变异分析的结果。
- Annotated Somatic Mutation(注释的体细胞突变):含详细注释的突变数据。
- Biospecimen Supplement(生物样本补充数据):关于生物样本的额外信息。
- Clinical Supplement(临床补充数据):患者的额外临床信息。
- Copy Number Segment(拷贝数片段):拷贝数变化信息。
- Gene Expression Quantification(基因表达定量数据):RNA-Seq等数据的表达量。
- Methylation Beta Value(甲基化Beta值):DNA甲基化数据。
- miRNA Expression Quantification(miRNA表达定量数据):miRNA表达信息。
- Pathology Report(病理报告):组织学分析的结果。
- Protein Expression Quantification(蛋白表达定量数据):蛋白组数据。
- Raw Intensities(原始信号强度):芯片数据的原始读数。
5. Data Format(数据格式)
存储数据的格式:
- bam:二进制对齐/映射格式
- bcr biotab:TCGA生物样本格式
- bcr omf xml:TCGA组织病理数据
- bcr pps xml:TCGA病理数据
- bcr ssf xml:TCGA患者随访数据
- bcr xml:TCGA整体数据存储格式
6. Workflow Type(数据处理流程)
表示数据的分析处理流程:
- ABSOLUTE LiftOver:基因拷贝数分析
- Aliquot Ensemble Somatic Variant Merging and Masking:体细胞突变合并和屏蔽
- Arriba:RNA融合分析工具
- ASCAT2/ASCAT3/AscatNGS:拷贝数变异分析工具
7. Platform(平台)
表示数据生成的平台,如Illumina、Affymetrix等。
8. Access(访问权限)
- Open Access:开放访问
- Controlled Access:受控访问(需要权限)
9. Tissue Type(组织类型)
- Primary Tumor:原发肿瘤
- Metastatic:转移瘤
- Normal Tissue:正常组织
10. Tumor Descriptor(肿瘤描述)
表示肿瘤的特征,如:
- Primary:原发肿瘤
- Recurrent:复发肿瘤
- Metastatic:转移性肿瘤
11. Specimen Type(标本类型)
- Fresh Tissue:新鲜组织
- FFPE(Formalin-Fixed Paraffin-Embedded):福尔马林固定石蜡包埋组织
12. Preservation Method(保存方式)
- Frozen:冷冻
- FFPE:石蜡包埋
为什么第一个筛选范围是从Experimental Strategy这里开始,而不是大家所熟知的,更加清晰的数据类别 Data Type(数据类型)开始?
这是因为 TCGA 数据库的筛选逻辑是基于数据生成过程的逻辑,而不是最终的数据存储结构。换句话说,TCGA 选择 Experimental Strategy(实验策略) 作为首要筛选项,是因为它决定了数据的来源和性质,而 Data Type(数据类型) 是实验数据进一步处理后的结果。
为什么从 Experimental Strategy(实验策略)开始?
-
实验策略决定了数据的本质,不同的实验策略会生成完全不同类型的数据。例如:
- RNA-Seq → 生成转录组数据(用于基因表达分析)
- WGS/WXS → 生成基因组变异数据(如 SNPs、CNVs)
- miRNA-Seq → 生成小RNA数据(miRNA 表达分析)
- ATAC-Seq → 研究染色质可及性
这些策略决定了数据的类型,而 Data Type 只是实验数据处理后的不同输出方式。
-
不同实验策略的数据存储格式、分析方法不同
例如,RNA-Seq 主要涉及 FPKM/TPM 格式的基因表达数据,而 WGS 主要涉及 BAM/SAM 格式的测序比对数据。如果直接从 Data Type(数据类型) 开始筛选,可能会导致不同实验策略的数据混杂在一起,不利于理解数据的来源和用途。 -
实验策略直接对应不同的研究需求
研究者通常从“想研究什么问题”出发:- 研究基因突变?选择 WGS / WXS
- 研究基因表达?选择 RNA-Seq
- 研究 DNA 甲基化?选择 Methylation Array
- 研究染色质开放性?选择 ATAC-Seq
这样,从实验策略出发,可以让研究者快速筛选适合自己研究需求的数据,而不是陷入各种数据类型的复杂选择中。
-
Data Type 是一个后续的“派生”分类
比如:- RNA-Seq 产生的 Gene Expression Quantification(基因表达定量)
- WGS 产生的 Simple Somatic Mutation(体细胞突变)
如果直接从 Data Type 开始筛选,可能会让研究者不清楚这些数据是如何生成的
我觉得一个直观的例子是 ,如果我们想研究基因突变,若从data category开始筛选,也许experimental strategy会包括了WGS、WXS两个实验方法,而事实上

WGS(全基因组测序)和 WXS(外显子组测序)的变异数据不等价
- WGS 检测的是 全基因组范围的变异(SNP、SV、CNV 等),包括编码区和非编码区。
- WXS 只检测 外显子区域的变异,无法发现调控区域的突变。
- 如果研究者误以为 WXS 能提供完整的基因组变异数据,可能会遗漏重要的非编码区突变。
或者说,如果我们想要研究最终的一个具体的特征是RNA转录水平,那么如果从data category开始选择,我们要选择的应该是transcriptome profiling,但这时候我们会发现experimental strategy留下了三个选项,也就是这三个实验方法都会生成data category是transcriptome profiling的数据,

而RNA-Seq 的基因表达数据 ≠ 基因芯片(Expression Array)的数据
- RNA-Seq 生成的是 TPM/FPKM/RPKM,数据是基于测序深度计算的。
- Expression Array 生成的是 荧光强度数据,本质是探针检测的信号。
- 如果混合使用 RNA-Seq 和 Expression Array 的基因表达数据,不经过标准化,可能会产生伪结果。

DAMO开发者矩阵,由阿里巴巴达摩院和中国互联网协会联合发起,致力于探讨最前沿的技术趋势与应用成果,搭建高质量的交流与分享平台,推动技术创新与产业应用链接,围绕“人工智能与新型计算”构建开放共享的开发者生态。
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容







所有评论(0)