从NCBI上快速下载目标物种基因组序列、获取SRA数据信息
从NCBI上快速下载目标物种基因组序列、获取SRA数据信息
·
1. 在genome 数据库中输入目标物种Genome List - Genome - NCBI (nih.gov)
输入目标物种
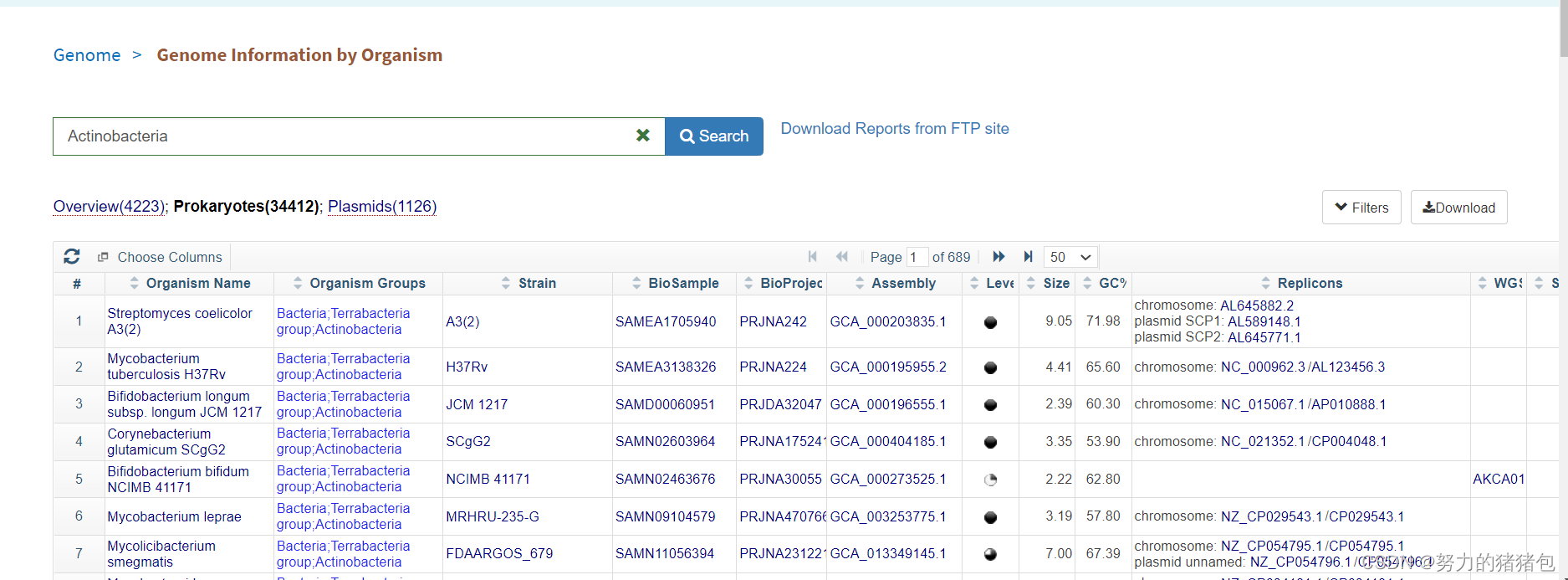
fliters 过滤,选择基因组装水平,宿主等
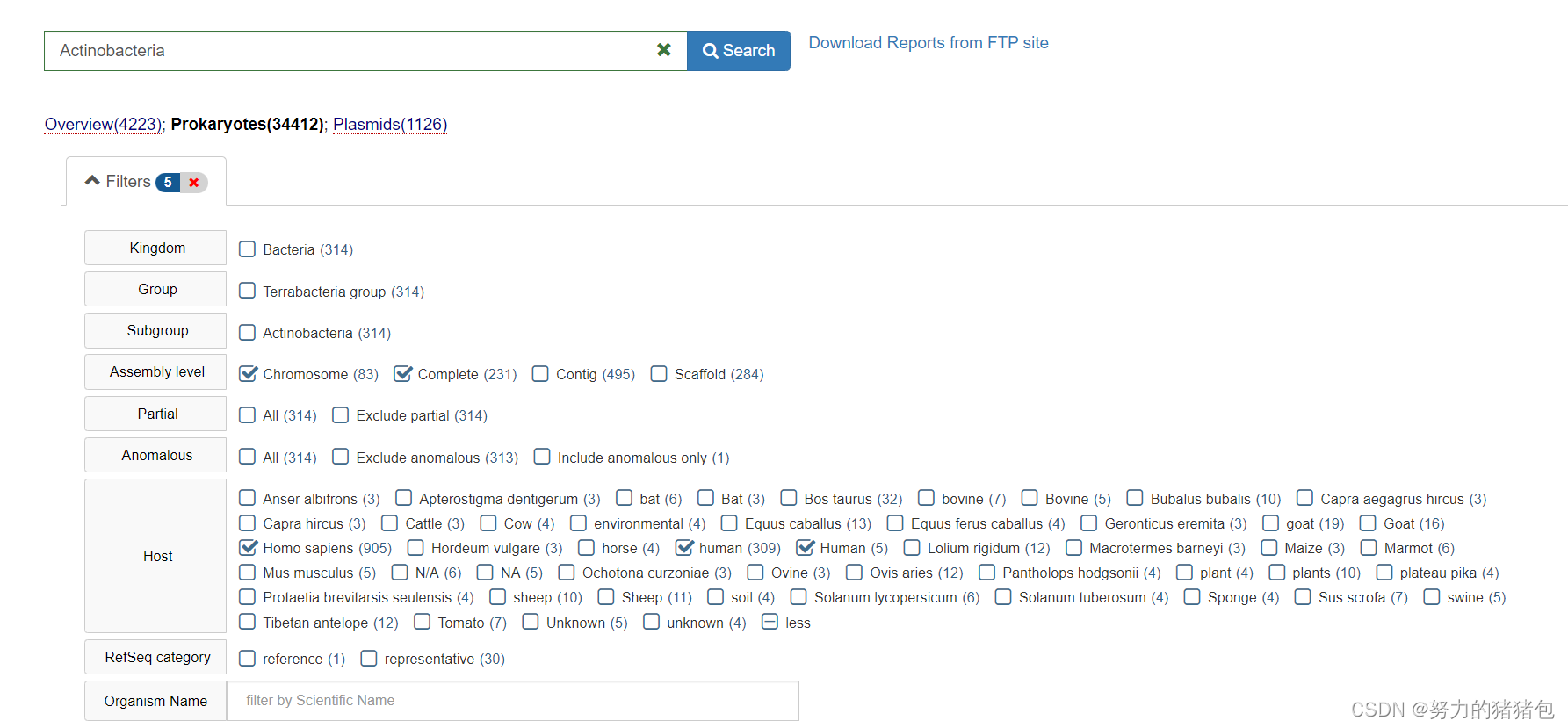 download 下载genome 数据结果——可获得基因组序列文件下载地址和对应的BioSample ID
download 下载genome 数据结果——可获得基因组序列文件下载地址和对应的BioSample ID

2. 提取 BioSample,另存在文本文件中

3. 使用NCBI Batch Entrez在BioSample数据库中下载相关信息得到SRA
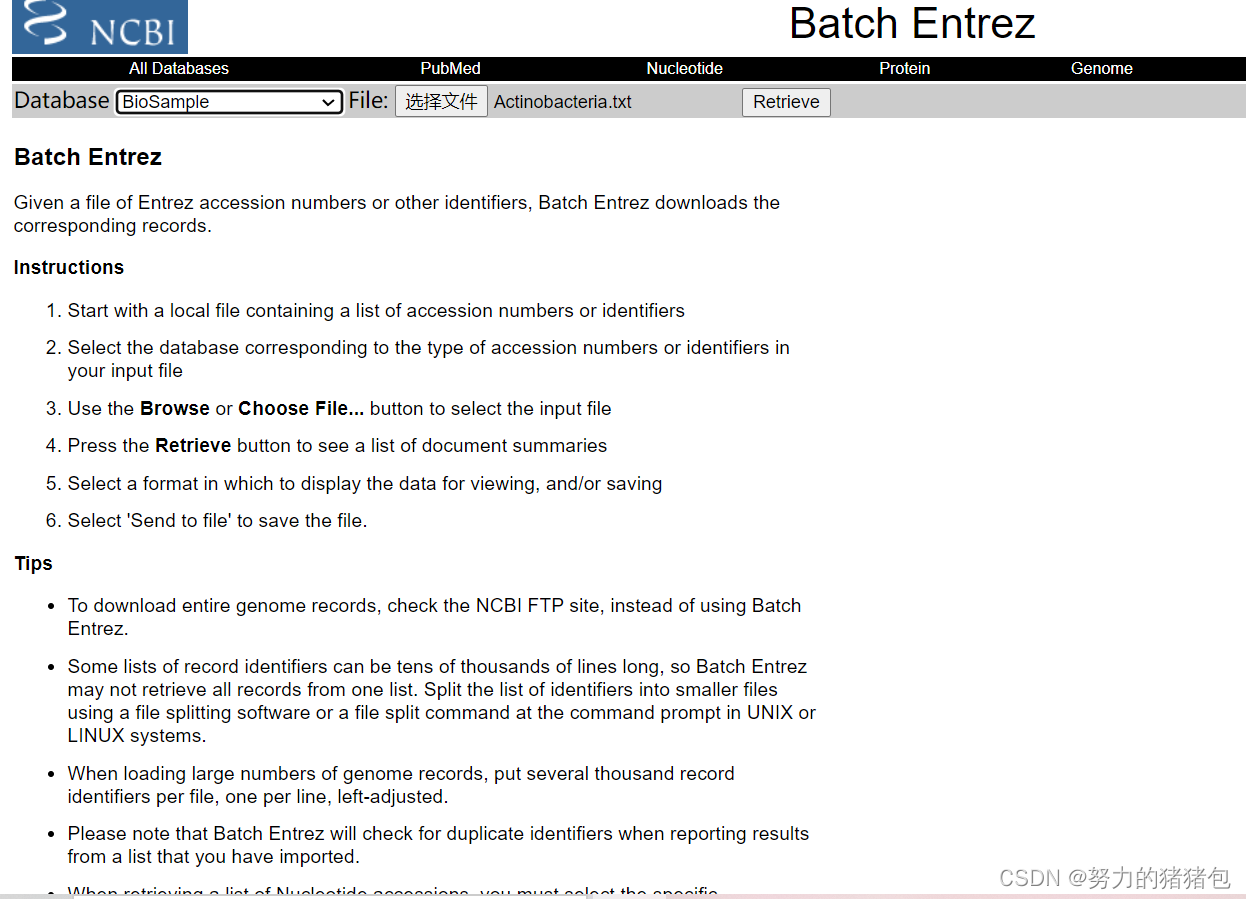
4. 在左边进行信息过滤,如过滤出能获取SRA文件的信息

send to 下载result,便可获得SRA信息

5. 写程序提取BioSample ,SRA ,Organism信息
import os
import pandas as pd
biosample=[]
organism=[]
sra=[]
filepath=r'E:\bacteria\Actinobacteria\biosample_result.txt'
with open ( filepath,'r') as file:
lines = file.readlines()
for line in lines:
if 'SRA:' in line:
sra.append(line.strip('\n').split('SRA:')[1].strip())
continue
if 'Organism:' in line:
organism.append(line.strip('\n').split('\t')[1])
continue
if 'Accession:' in line:
biosample.append(line.strip('\n').split('Accession:')[1].split('\t')[0].strip())
continue
outpath = r'E:\bacteria\Actinobacteria\Actinobacteria.xlsx'
df=pd.DataFrame({'BioSample':biosample,'Phylum':'Actinobacteria','Organism':organism,'SRA':sra})
df.to_excel(outpath,index=False,header=False)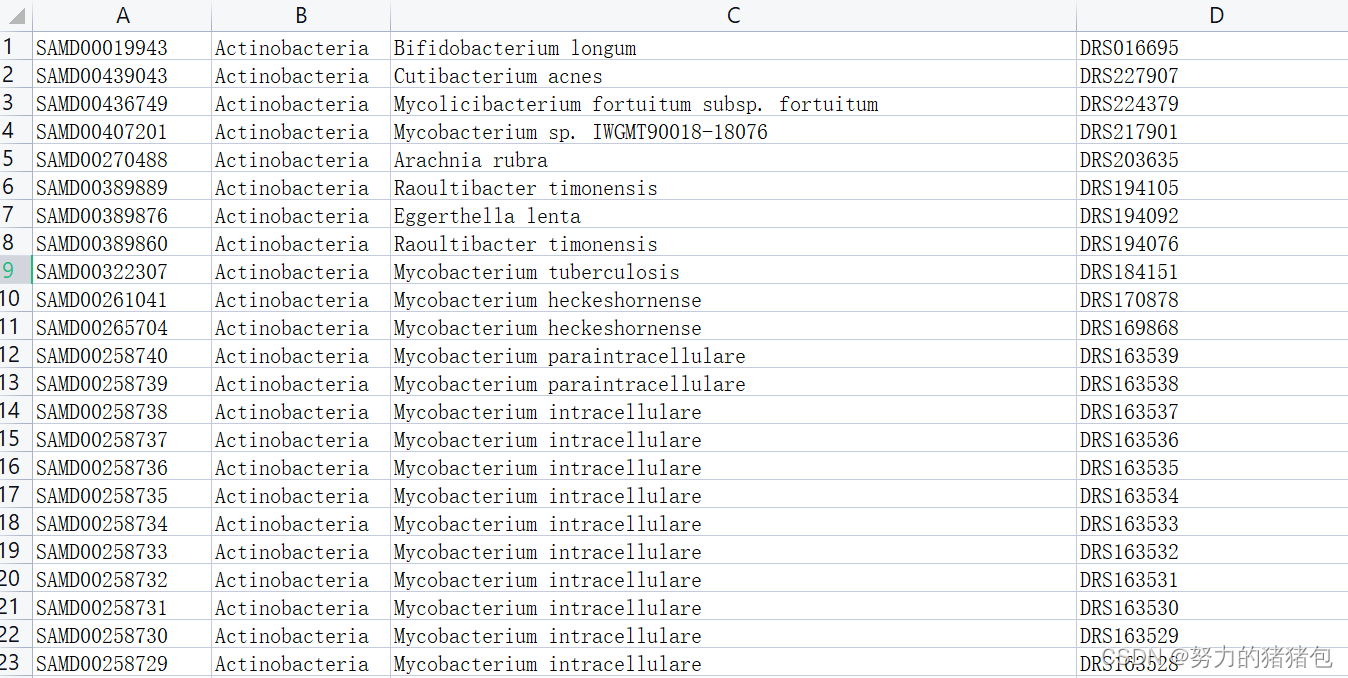
6. 将organism信息去重复后另存为文本文件

7. 使用NCBI Batch Entrez在Taxonomy数据库中获取lineage


DAMO开发者矩阵,由阿里巴巴达摩院和中国互联网协会联合发起,致力于探讨最前沿的技术趋势与应用成果,搭建高质量的交流与分享平台,推动技术创新与产业应用链接,围绕“人工智能与新型计算”构建开放共享的开发者生态。
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容








所有评论(0)