MPB | 宁波大学王凯等-海洋环境资源状态划分及微生物群落生态位宽度计算方法...
海洋环境资源状态划分及微生物群落生态位宽度计算方法Partitioning of Resource States in the Marine Environment and Calculation of Niche Breadth of Microbial Community 闫慧贞,张德民,王凯*海洋学院,宁波大学,宁波市,浙江省*通讯作者邮箱: wangkai@nbu.edu.cn引用格式:闫
海洋环境资源状态划分及微生物群落生态位宽度计算方法
Partitioning of Resource States in the Marine Environment and Calculation of Niche Breadth of Microbial Community
闫慧贞,张德民,王凯*
海洋学院,宁波大学,宁波市,浙江省
*通讯作者邮箱: wangkai@nbu.edu.cn
引用格式:闫慧贞,张德民,王凯. (2022). 海洋环境资源状态划分及微生物群落生态位宽度计算方法. Bio-101 e2003391. Doi: 10.21769/BioProtoc.2003391.
How to cite: Yan, H. Z., Zhang, D. M. and Wang, K. (2022). Partitioning of Resource States in the Marine Environment and Calculation of Niche Breadth of Microbial Community. Bio-101 e2003391. Doi: 10.21769/BioProtoc. 2003391. (in Chinese)
摘要:生态位宽度 (niche breadth) 是衡量群落中物种环境适应度的重要指标。近年,经典Levins生态位宽度指数开始被用于表征微生物的环境适应度,在计算该指数时首先要评估样地资源状态数量,但在高度连通且复杂多变的海洋环境中评估资源状态数难度较大。本教程提出用非层次聚类分析综合多项海水理化因子对研究海域资源状态进行划分,随后计算Levins生态位宽度。该方法能较客观地评估海区环境异质性,从而避免简化地将单个样点视作一个资源状态,进而避免因高估样地资源状态数而导致的计算偏差。此外,该方法还降低了资源状态指标的维度,从而降低了生态位宽度算法的复杂度。
关键词: 非层次聚类,资源状态,生态位宽度,微生物群落
仪器设备
1.个人电脑(安装Windows 10操作系统)
软件
1. R (v3.5.0)
2. Microsoft Office Excel (v2016)
实验步骤
生态位宽度 (niche breadth) 能反映出不同物种对各种环境条件的适应能力差异。生态位宽度可依据非生物或生物变量(如最适生长温度、氧亲和力、营养类型等)进行量化,前提是准确定义及划分生境资源状态 (resource states)。资源状态可从多种角度被定义,如食物资源(食物的种类或尺寸)、栖息地资源(理化环境因子)或自然采样单元 (Krebs, 2014)。本教程采用研究区域的理化环境因子综合评估资源状态。在计算海洋微生物群落的生态位宽度时,首先要准确评估研究区域的资源状态数量,但在高度连通且复杂多变的海洋环境中评估资源状态数难度较大。本教程提出用非层次聚类分析综合多项海水理化因子对研究海域资源状态进行划分,随后计算Levins生态位宽度。操作步骤如下:
1.示例数据来源
本教程描述的海洋环境资源状态划分方法由本课题组提出并应用 (Wang等,2020),因此以该论文中的部分数据作为示例数据,随后的生态位宽度计算采用经典Levins算法 (Levins 1968)。示例数据可从Github获取:https://github.com/huizhen-yan/BioProtocol-NicheBreadth。
2.基于非层次聚类划分海洋环境资源状态
本教程将海洋环境资源状态定义为海水环境条件的最优分类 (optimal categories)。用R语言软件包vegan (Oksanen等,2018) 中的函数cascadeKM()调用k-均值划分法 (k-means partitioning)对海水环境因子(图1)进行非层次聚类,用SSI指数 (Simple Structure Index) 确定最佳分组数,最大的SSI值表示最佳聚类方式,其对应的分组数即为研究区域中所有采样站位的资源状态数。所用R代码如图2所示,用函数plot()绘制k-均值聚类结果(图3),表明示例数据的最佳分组数为8;所有采样站位的具体分组情况如图4所示,根据"8 groups"一列即可确定每个样本所属的组别。

图1. 海水环境因子表(局部)

图2. 基于k-均值划分样地资源状态的R代码

图3. 在R环境中导出k-均值聚类图 (Wang等,2020)
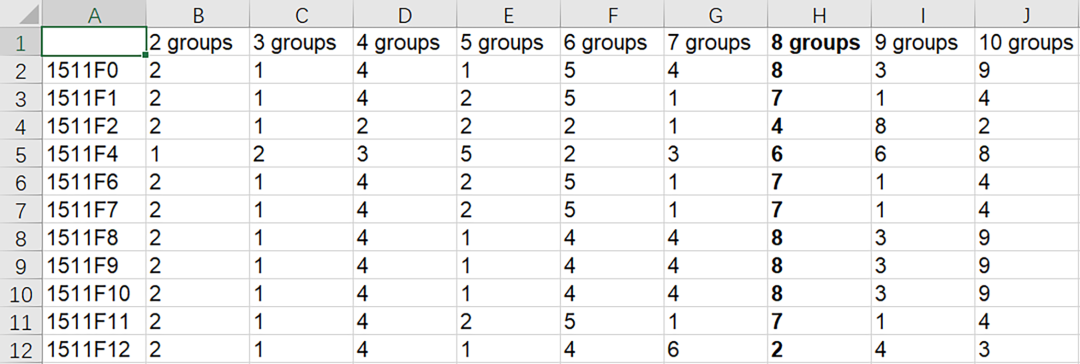
图4. k-均值聚类结果 (env.KM.partition表). 注:加粗代表最佳分组方式
3.将微生物OTU表转化为微生物-资源状态分布表
按照上一步得到海区站位资源状态分布情况,把属于同一资源状态的所有站位中各物种丰度分别求和,即为该资源状态下的物种分布情况。在Excel中将微生物OTU(Operational Taxonomic Unit,分类操作单元)表转换为OTUs在各资源状态中的分布表(图5)。

图5. 微生物-资源状态分布表 (RSMatrix.txt)
4.生态位宽度计算
Levins (1968) 提出通过计算各资源状态中某物种分布的均匀度来评估该物种的生态位宽度。Levins生态位宽度 (B) 的计算公式为:

其中Bj表示物种j的生态位宽度,Pij表示物种j在资源状态i中的比例,N表示资源状态总数,Bj的取值范围是1-N。由于不同采样时间点(如不同季节)海洋环境资源状态总数往往不同,导致Bj的范围不同,为了便于比较,有必要将Bj标准化至统一数值范围(0-1) (Hurlbert 1978)。标准化Levins生态位宽度 (BA) 的计算公式为:


图6. 计算物种水平生态位宽度的R代码
计算得到每个物种的标准化生态位宽度BA后(图6),单个样本中所有物种的生态位宽度BA的平均值即为该样本群落水平的生态位宽度Bcom (Wu等,2018)。群落水平的生态位宽度Bcom计算R代码如下(图7):

图7. 计算群落水平生态位宽度的R代码
致谢
本项工作受国家自然科学基金 (项目编号:41977192)、宁波市自然科学基金 (项目编号:2021J060)、宁波大学省属高校基本科研业务费项目 (项目编号:SJLY2022001) 的资助。
作者贡献声明:王凯和闫慧贞提出该方法的应用思路,闫慧贞实现该方法,闫慧贞和王凯撰写文章,张德民审阅并修改文章。
参考文献
1.Krebs, C. J. (2014). Ecological methodology-3rd Version, pp.597-605. Addison-Welsey Educational Publishers, Inc., Menlo Park, CA.
2.Wang, K., Yan, H. Z., Peng, X., Hu, H. J., Zhang, H. J., Hou, D. D., Chen, W., Qian, P., Liu, J. F., Cai, J. B., Chai, X. L. and Zhang, D. (2020). Community assembly of bacteria and archaea in coastal waters governed by contrasting mechanisms: a seasonal perspective. Molecular Ecology 29(19): 3762-3776.
3.Levins, R. (1968). Evolution in changing environments. Princeton University Press, Princeton, New Jersey.
4.Oksanen, J., Blanchet, F. G., Friendly, M., Kindt, R., Legendre, P., McGlinn, D., Minchin, P. R., O'Hara, R. B., Simpson, G. L., Solymos, P., Stevens, M. H. H., Szoecs, E. and Wagner, H. (2018). vegan: Community Ecology Package. R package version 2.5-6. Available from http://CRAN.r-project.org/package=vegan.
5.Hurlbert, S. H. (1978). The measurement of niche overlap and some relatives. Ecology 59(1): 67-77.
6.Wu, W. X., Lu, H. P., Sastri, A., Yeh, Y. C., Gong, G. C., Chou, W. C., Hsieh, C. H. (2018). Contrasting the relative importance of species sorting and dispersal limitation in shaping marine bacterial versus protist communities. The ISME Journal 12(2): 485-494.
宏基因组推荐
本公众号现全面开放投稿,希望文章作者讲出自己的科研故事,分享论文的精华与亮点。投稿请联系小编(微信号:yongxinliu 或 meta-genomics)
猜你喜欢
iMeta高引文章 fastp 复杂热图 ggtree 绘图imageGP 网络iNAP
iMeta网页工具 代谢组MetOrigin 美吉云乳酸化预测DeepKla
iMeta综述 肠菌菌群 植物菌群 口腔菌群 蛋白质结构预测
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature
一文读懂:宏基因组 寄生虫益处 进化树 必备技能:提问 搜索 Endnote
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流快速解决科研困难,我们建立了“宏基因组”讨论群,己有国内外6000+ 科研人员加入。请添加主编微信meta-genomics带你入群,务必备注“姓名-单位-研究方向-职称/年级”。高级职称请注明身份,另有海内外微生物PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。
点击阅读原文

DAMO开发者矩阵,由阿里巴巴达摩院和中国互联网协会联合发起,致力于探讨最前沿的技术趋势与应用成果,搭建高质量的交流与分享平台,推动技术创新与产业应用链接,围绕“人工智能与新型计算”构建开放共享的开发者生态。
更多推荐
 已为社区贡献47条内容
已为社区贡献47条内容





所有评论(0)